
Omics Analysis with Apache Spark 第2回 GTEx eQTLデータ編
前回はGTEx eQTLのAdipose_SubcutaneousをApache SparkのDataFrameに取り込みました。今回はいよいよそれを全tissueに拡大します。また、容量も大きくなってきましたので今回はmacではなくAWS上に構築したSpark Clusterを使用します。
データの下準備
GTExのデータはgzipされていますがApache Sparkで分散処理を行うのにあたりbgzipに変換します。これで分散性能がかなり変わってきます。
gsutilでファイルを取得 -> gunzip -> bgzip -> s3へ配置
という流れで前準備を行っています。
起動
あらかじめ設定しておいたAWSの"Service Catalog"からパラメータを入力し、欲しいサイズのSpark Clusterを起動していきます。
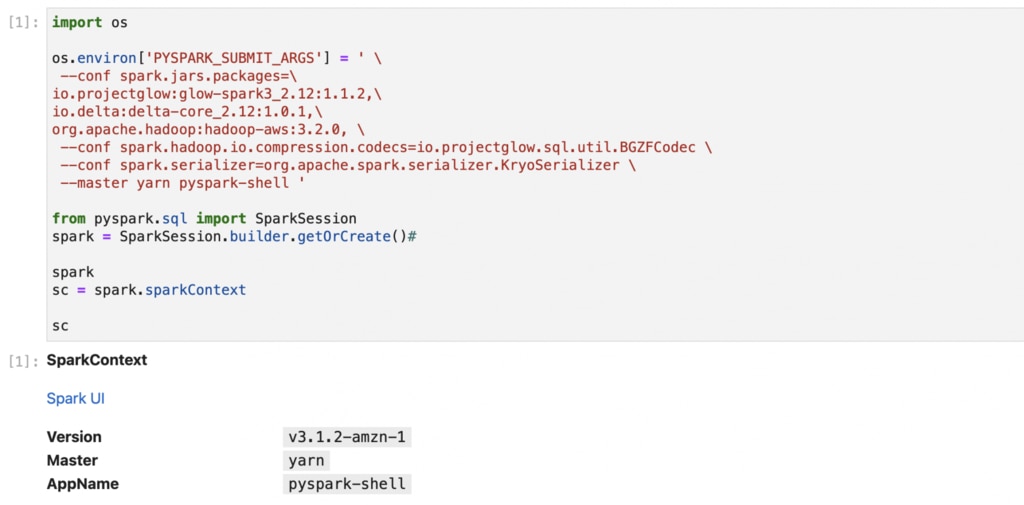
今回の処理に必要最低限なオプションは以下のとおりです。
| オプション | 役割等 |
|---|---|
| --packages | packages以下にコンマ区切りで依存関係にあるパッケージを指定しています。 |
| io.projectglow:glow-spark3_2.12:1.1.2, io.delta:delta-core_2.12:1.0.1, |
この2行はdeltaを使うために必要です。 |
| org.apache.hadoop:hadoop-aws:3.2.0, | s3へアクセスするために必要なものです。 |
| --conf spark.driver.memory=12G | driverに12GBのメモリを割り当てています。 今回はClusterではないのですがその場合はdriverがexecutorの役割を担うためこのようにしています。 |
| --conf spark.serializer=org.apache.spark.serializer.KryoSerializer | serializeにつかうclassを指定。推奨値です。 |
データの変換
前回と同様に、variant_idは chr1_13550_G_A_b38のように複数の情報がひとつになっていてこのままでは利活用に支障があると思われるため、次のような列に変換をすることにします。
また、後で他のtissueも結合しますので新たに「tissue」を追加します。
スキーマも前回と同じものを使えます。
| 列名 | 内容 |
|---|---|
| contigName | chr1 |
| start | 13550 |
| referennceAllele | G |
| alternateAlleles | A |
| tissue | Adipose_Subcutaneous |
以降で使うモジュールをロードし、ファイル名とtissueを定義します。

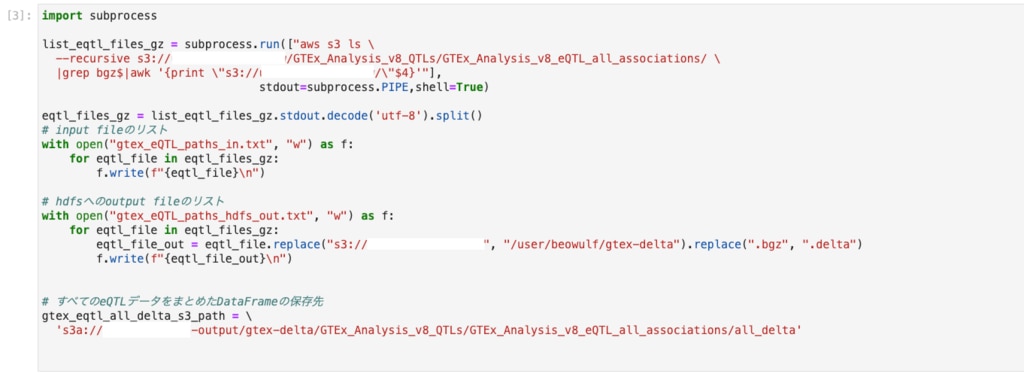
ファイルのリストから、inputファイルのリストやoutputファイルのリストを作成しておきます。
今回はs3とhdfsの両方に出力していますが本来はどちらか一方でよいと思います。
処理の内容で中間ファイルとして集めるものが多ければhdfsの方が高速ですし、
そういった要素が少なく、最後に結果を保存するのみであればs3だけでよさそうです。
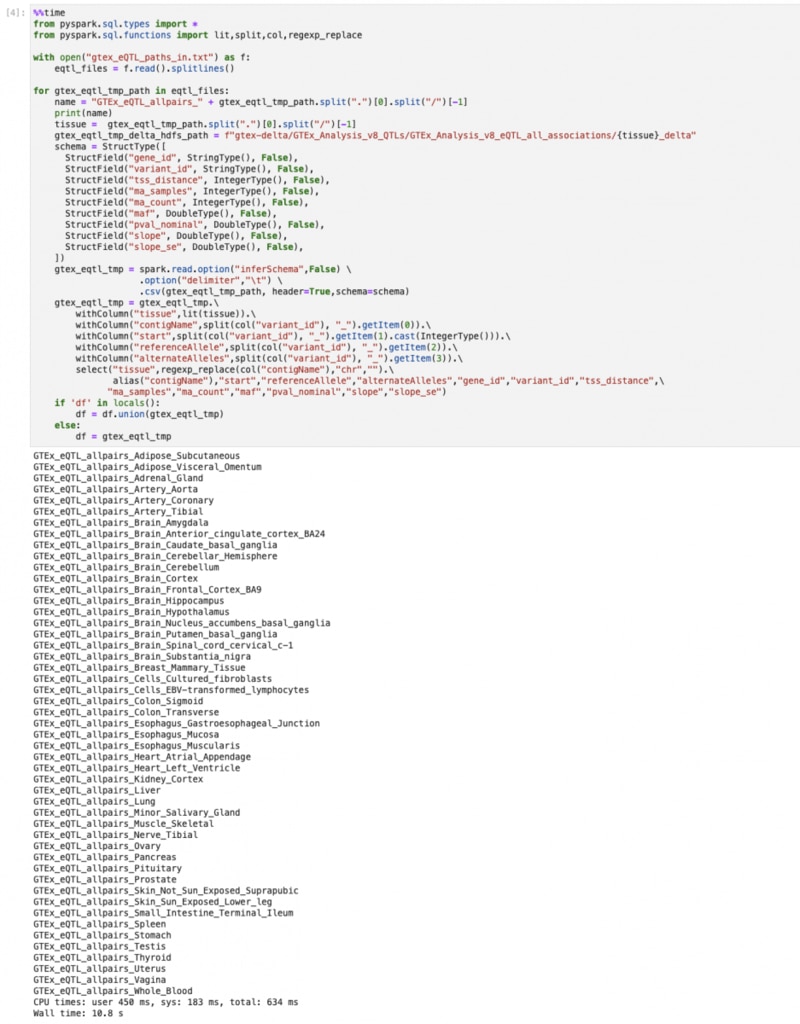
リストをもとにbgzファイルを読み込み、前回つくったSchemaを使ってDataFrameに変換します。
同時に、sparkのunionでつなげていき、1つの大きなDataFrameにしています。
個々のtissueのDataFrameをここでs3へ保存してもよいのですが、せっかくSparkによる大規模データ処理を想定しているので今回は必要なしとします。
ディスクへの書き込み、読み込み
S3
結合したeQTLデータをdelta形式でs3へ保存します

s3から読み込む場合は、このようにします。

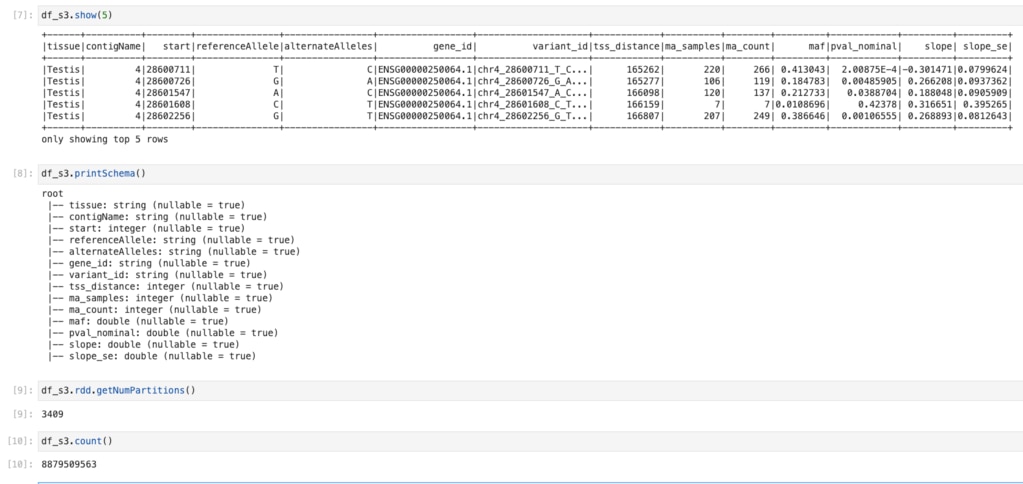
読み込んだDataFrameを確認します
DataFrame操作
読み込んだDataFrameは次のようにFilterなどの操作ができます。
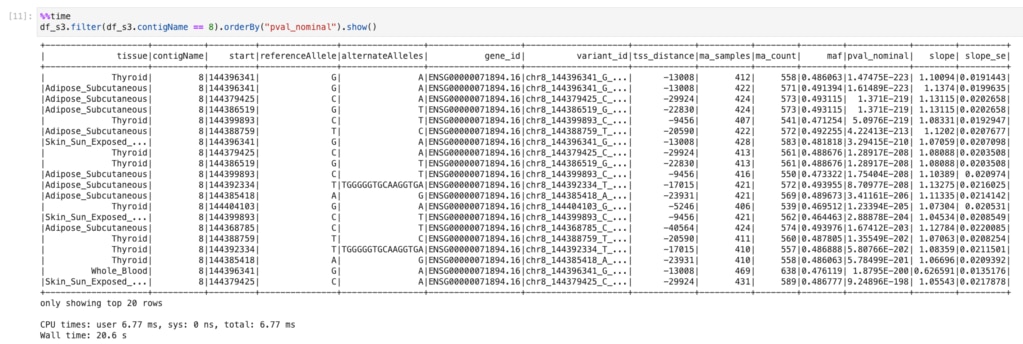
ここでは8番染色体のみに絞り込み、pval_nominalをsortしています。
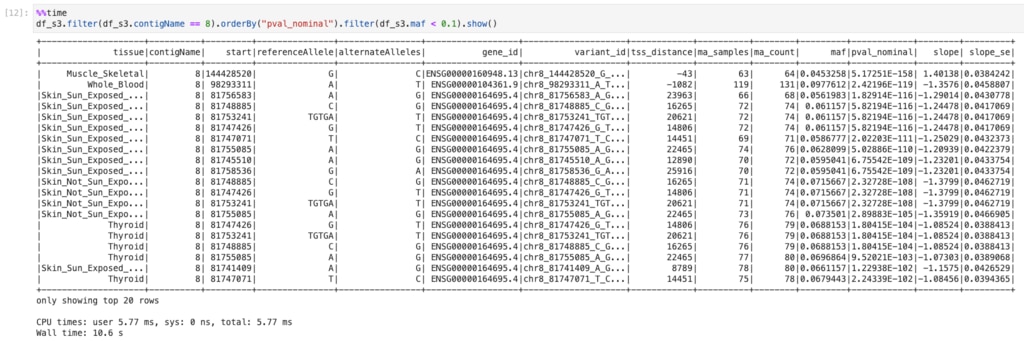
filterを追加してmaf < 0.1のみを表示させてみます。
GTExのeQTL データを全tissue分DataFrameにし、s3へ保存、DataFrameの簡単な操作をしてみました。前半の部分はデータの更新時にのみ行うようなものでそれほど頻度は高くないと思います。一度s3へ保存してしまえば、すぐにSparkのDataFrameとして読み込み、分析に使えます。 Cloudを使ったSpark Clusterなのでインタラクティブな操作で素早いレスポンスが欲しい場合はノードを多数使ってもよいですし、スクリプトによるバッチ処理でゆっくり結果が出てくればよいのであれば相応のインスタンスを使ってコストパフォーマンスを出してもよいという柔軟性が魅力です。 今回はc5.9xlargeを16台用い、テスト用でしたのですべてSPOTインスタンスを使いました。 次回はsQTLのデータをDataFrameに読み込んでみようと思います。

