
Omics Analysis with Apache Spark 第3回 GTEx sQTLデータ編
今回はGTEx sQTLのAdipose_SubcutaneousをApache SparkのDataFrameに取り込みます。容量も大きいのでAWS上に構築したSpark Clusterを使用します。
データの下準備
GTExのsQTLデータはparquet形式で保存されていましたので、そのままApache SparkのDataFrameへ読み込むことができます。
あらかじめダウンロードしてs3へ置いておきます。
起動
あらかじめ設定しておいたAWSの"Service Catalog"からパラメータを入力し、欲しいサイズのSpark Clusterを起動していきます。
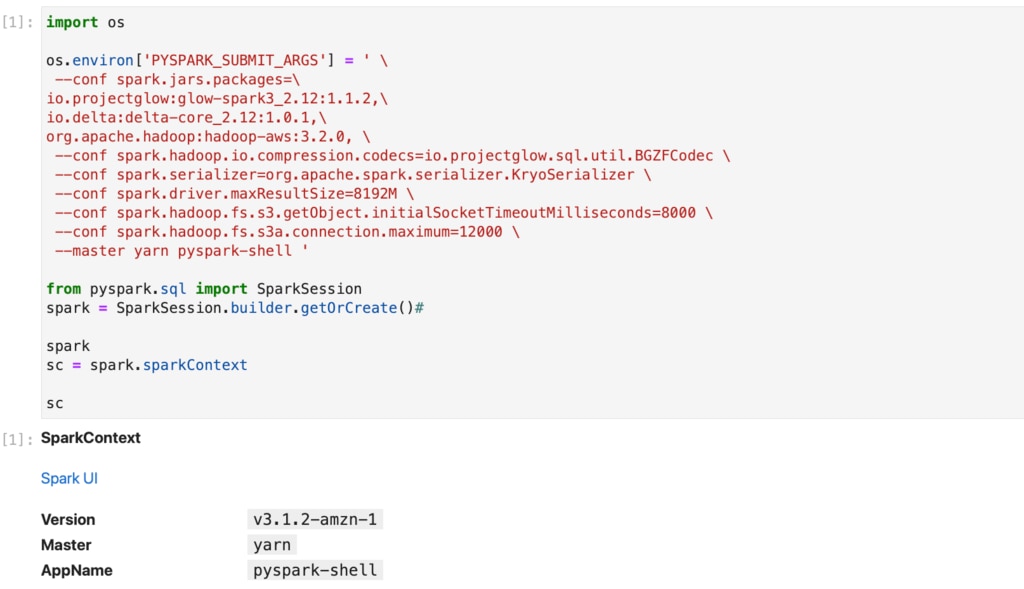
今回の処理に必要最低限なオプションは以下のとおりです。
| オプション | 役割等 |
|---|---|
| --packages | packages以下にコンマ区切りで依存関係にあるパッケージを指定しています。 |
| io.projectglow:glow-spark3_2.12:1.1.2, io.delta:delta-core_2.12:1.0.1, |
この2行はdeltaを使うために必要です。 |
| org.apache.hadoop:hadoop-a | s3へアクセスするために必要なものです。 |
| --conf spark.serializer=org.apache.spark.serializer.KryoSerializer | serializeにつかうclassを指定。推奨値です。 |
| --conf spark.driver.maxResultSize=8192M \ --conf spark.hadoop.fs.s3.getObject.initialSocketTimeoutMilliseconds=8000 \ --conf spark.hadoop.fs.s3a.connection.maximum=12000 |
今回のデータ処理を完遂するために必要だったオプションです。 |
データの変換
前回と同様に、variant_idは chr1_13550_G_A_b38のように複数の情報がひとつになっていてこのままでは利活用に支障があると思われるため、次のような列に変換をすることにします。
また、後で他のtissueも結合しますので新たに「tissue」を追加します。
スキーマも前回と同じものを使えます。
| 列名 | 内容 |
|---|---|
| contigName | chr1 |
| start | 13550 |
| referennceAllele | G |
| alternateAlleles | A |
| tissue | Adipose_Subcutaneous |
以降で使うモジュールをロードし、ファイル名とtissueを定義します。

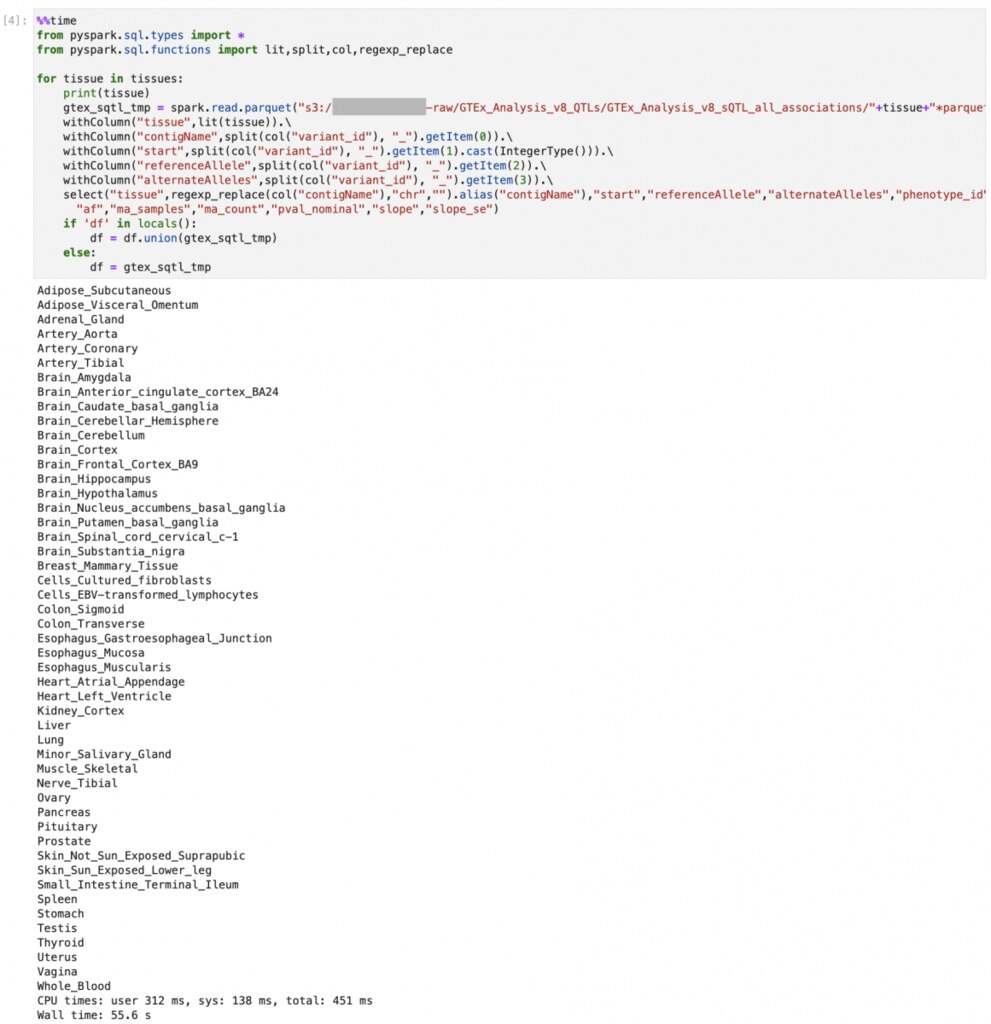
ファイルのリストから、tissueのリストをつくっておきます。リストをもとにparquetファイルを読み込み、DataFrameに変換します。
同時に、sparkのunionでつなげていき、1つの大きなDataFrameにしています。

リストをもとにparquetファイルを読み込み、DataFrameに変換します。
同時に、sparkのunionでつなげていき、1つの大きなDataFrameにしています。
ディスクへの書き込み、読み込み
S3
結合したsQTLデータをdelta形式でs3へ保存します

s3から読み込む場合は、このようにします。

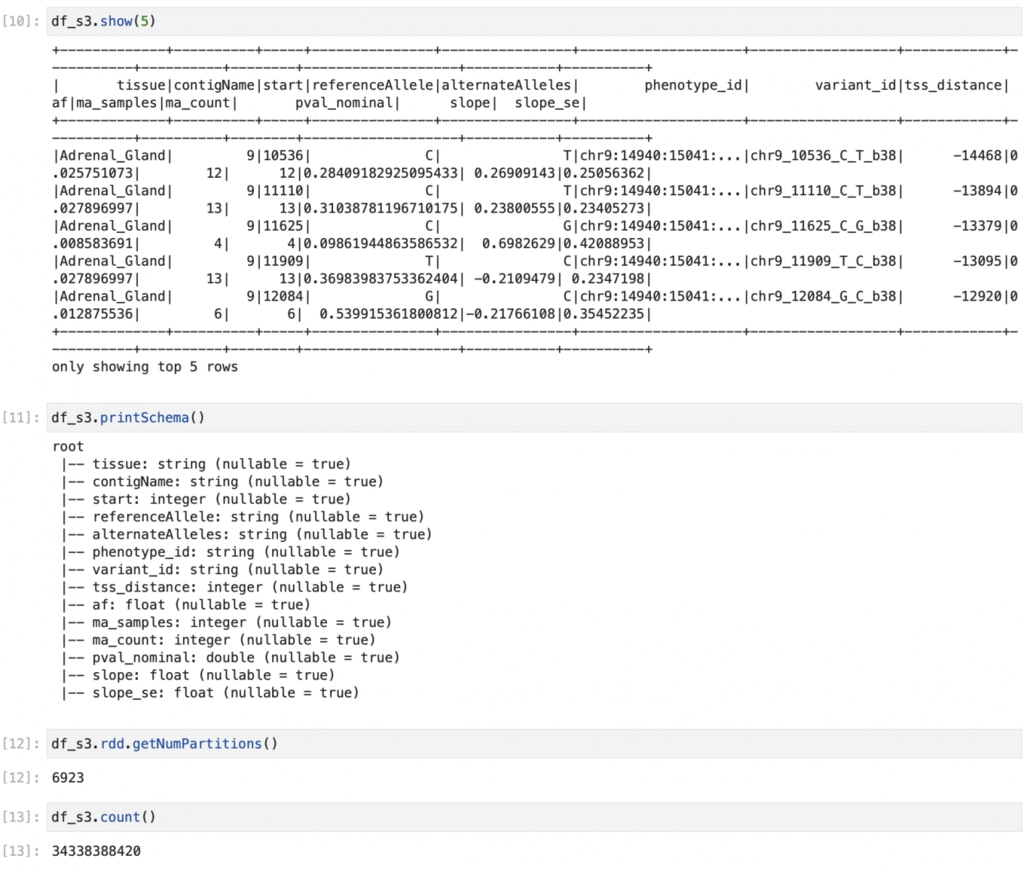
読み込んだDataFrameを確認します
DataFrame操作
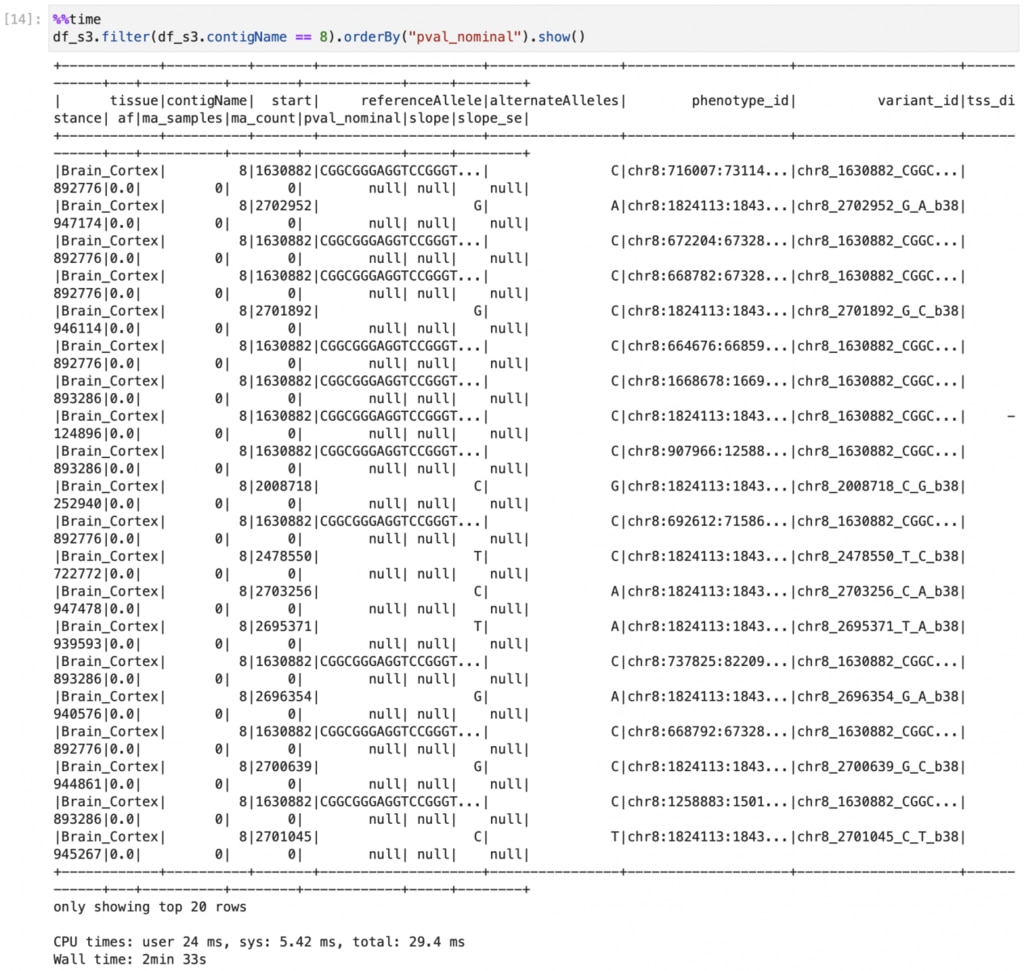
読み込んだDataFrameは次のようにFilterなどの操作ができます。
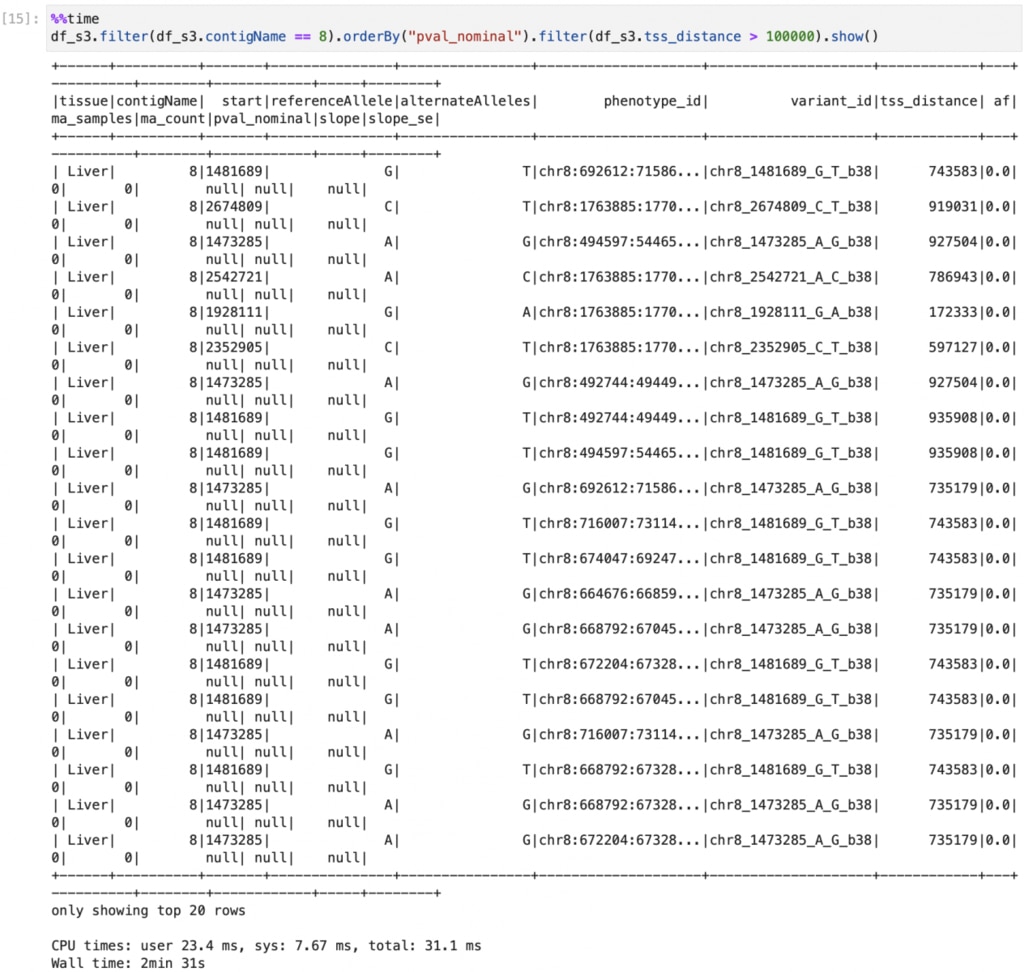
ここでは8番染色体のみに絞り込み、pval_nominalをsortしています。
filterを追加してtss_distance >100000のみを表示させてみます。
今回までで、GTExのeQTLおよびsQTLのデータをSparkのDataFrameへ読み込みました。eQTLのときはgzファイルでしたが、sQTLはparquetファイルでした。Apache Sparkはかなり多様なデータの読み込みが可能です。一度DataFrameにしてdelta形式で保存しておけばいつでもSparkから呼び出して参照することができます。 次回は未定ですが、また何か別のデータを読み込んでみようと思います!

