Galaxyをわかりたい! (6) APIを使ったGalaxyへのアクセス
前回までの記事 Galaxyをわかりたい!(1) Galaxyのキホン Galaxyをわかりたい!(2) Galaxyの拡張機能紹介 Galaxyをわかりたい!(3) Dockerイメージをカスタマイズして使えるPythonやRStudio Galaxyをわかりたい!(4) Galaxyでのユーザー管理について Galaxyをわかりたい!(5) Galaxy内部の動き 今回は、「Galaxyをわかりたい!(2) Galaxyの拡張機能紹介」でも少しふれた、Galaxy APIにアクセスするためのライブラリBioblendをご紹介します。Bioblendを使うとpythonで自由度の高い操作ができます。
インストール
Galaxy本体もpythonで書かれており、インストール時にBioblendのライブラリも一緒にインストールされます。virtualenvでGalaxyのpython環境をロードすると使えるようになります。 手元のPCなど、Galaxyがインストールされていない環境で使用するときは、pipでbioblendのみインストールします。 https://bioblend.readthedocs.io/en/stable/#installation実行例
Galaxyサーバーへの接続
GalaxyのURLとユーザーのAPI keyで操作するインスタンスを指定します。上記の例はローカルホストを指定していますが、リモートホストのインスタンスを指定することもできます。アクセスするユーザーの権限により実行可能な操作が違っており、たとえばデータライブラリを作成できるのは管理者権限のあるユーザーのAPI keyを使用したときのみです。API keyはweUIのユーザー設定から作成することができます。ヒストリーにデータセットをアップロードする
接続先とユーザーのAPI keyを指定し、データセットをアップロードします。ここでは、リモートのクライアントからデータセットをアップロードしています。Galaxyの実行ホストにログインする必要はありません。 アップロードしたデータセットのhidを確認します。hidはヒストリーの各データセットごとに表示される番号です。 webUIから確認します。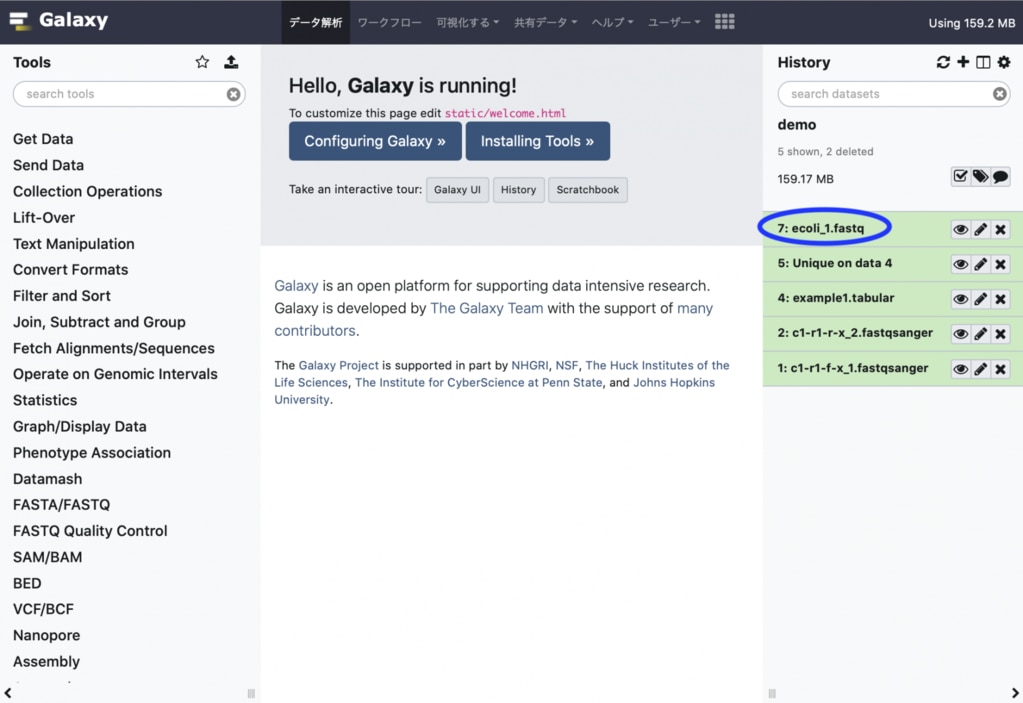 pythonコンソールからアップロードしたものと同じ名前のファイルがヒストリーに表示されています。hidが同じ7となっていることがわかります。
pythonコンソールからアップロードしたものと同じ名前のファイルがヒストリーに表示されています。hidが同じ7となっていることがわかります。
ライブラリを作成してデータをアップロードする
"ProjectA_library"という名前で新規データライブラリを作成します。 データセットをアップロードします。ここでは、ホストのローカルファイルシステムにあるファイル群をアップロードしています。データライブラリインポート用に設定したディレクトリ にあらかじめファイルを配置しておく必要があります。 webUIから確認します。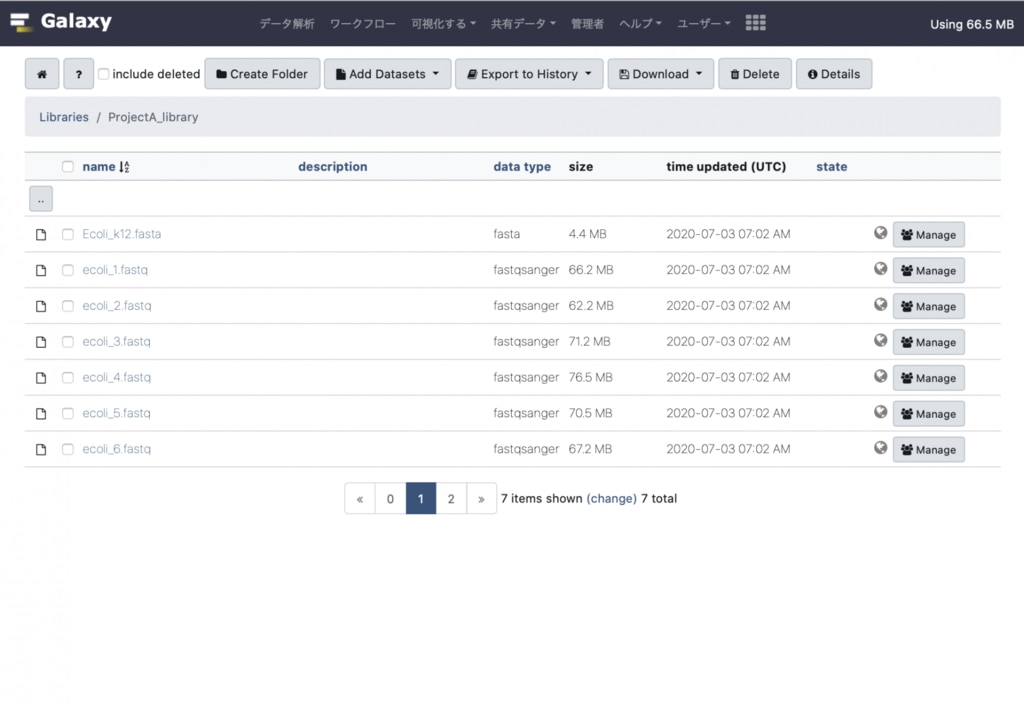 ProjectA_libraryに複数のデータがアップロードされています。
ProjectA_libraryに複数のデータがアップロードされています。