Galaxyをわかりたい!(1) Galaxyのキホン
webベースのバイオメディカル解析プラットフォームとしてもはや定番と言ってもいいGalaxyですが、「名前は聞いたことあるけれど使ったことはない。」「便利らしいけれどどう使うのかわからない。」という方も多いと思います。ここで基本に立ち返って、Galaxyとは何なのか、大まかにご紹介します。
Galaxyの生い立ち
Galaxyは2005年に開発がスタートし、2007年に公共サーバーが公開されました。以降Penn State University, Johns Hopkins university , Oregon Health & Science University, Cleveland Clinicの共同コミュニティを中心にオープンソースで開発が続けられています。また開発者やユーザーが参加するカンファレンスやワークショップが世界各地で行われています。 Galaxyにはツールを追加するためのToolShedという仕組みがあり、GalaxyにおけるAppStoreとも言えるものです。開発元のToolShedでは、2019年9月の時点で 7354のツールが配布されています。これらのツールは開発者が自由に公開でき、ローカルやクラウドの自前Galaxyの管理者は自由にインストールできます。各地の開発者がさまざまなツールを作ることで、対応ツールの増加を加速させていると言えるでしょう。本家の公共Galaxy
Galaxy開発元の公共サーバーです。インターネット経由で誰でも無料で使うことができます。ユーザーアカウントを作らずに使うこともできますが、ワークフローの機能を使うにはアカウントが必要です。またアカウントを作ることで、別の場所からでも持続した解析を行うことができます。 https://usegalaxy.org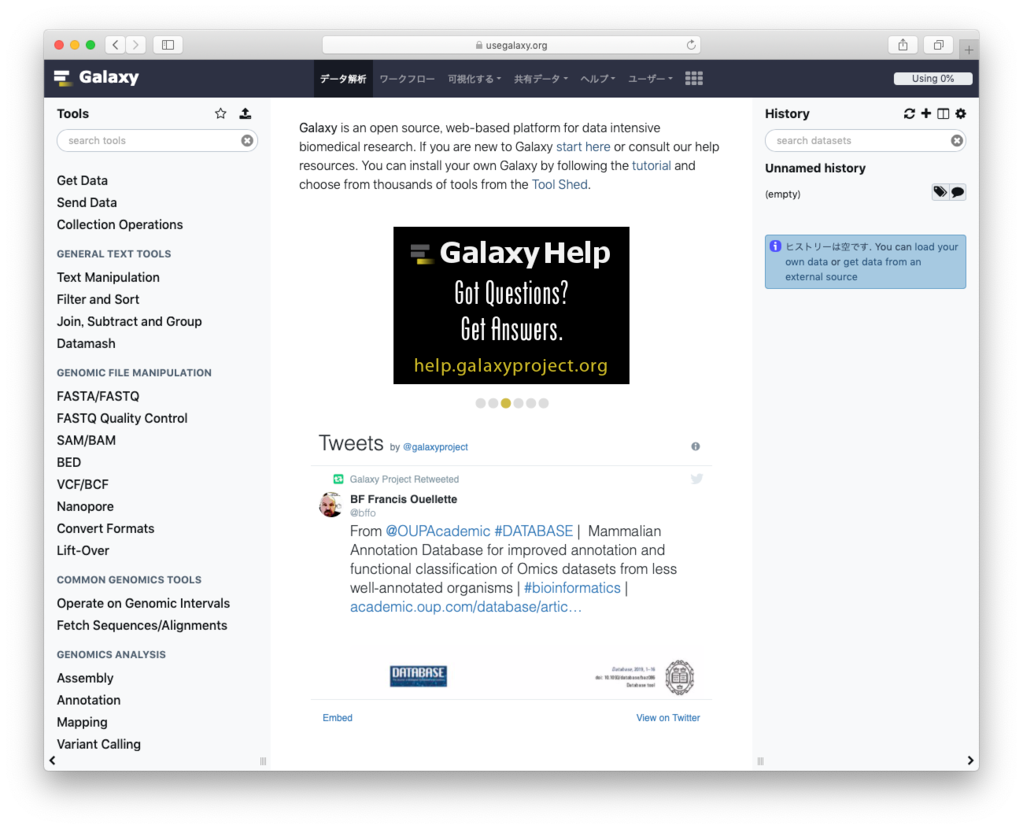 基本的には以下のような流れで解析を行います。
基本的には以下のような流れで解析を行います。
-
- データを右側のヒストリーにアップロードする
- 左側のツールパネルからツールを選ぶ
- 中央のパネルで入力データやパラメータを選ぶ
- ジョブを実行
- 実行結果が右側のヒストリーに表示される
その他の公共Galaxy(地域別、目的別)
開発元が公開しているものの他にも多くのサーバーが公開されています。基本的な使い方はいずれも同じですが、サイトによってはユーザー登録が必要な場合があります。 Galaxy Platform Directoryで目的別に探すことができます。 https://galaxyproject.org/use/ ツールが多くインストールされているUseGalaxyは、本家の他にヨーロッパ、オーストラリアで公開されています。 https://usegalaxy.eu (Europe) https://usegalaxy.org.au (Australia)
https://usegalaxy.org.au (Australia)
 そのほか、ある解析に特化したGalaxyも多数公開されています。ツールが絞られているかわりに、UseGalaxyには入っていないツールや独自に開発されたツールがインストールされています。以下がそのようなサイトの例です。
最近の動向としては、EBIがシングルセル向けのGalaxyツールを複数開発し下記のHuman Cell Atlas Galaxyに乗せています。
https://galaxyproject.org/use/human-cell-atlas/ (シングルセル解析に特化)
https://hicexplorer.usegalaxy.eu (Hi-C解析に特化)
http://huttenhower.sph.harvard.edu/galaxy/ (メタゲノム解析に特化)
そのほか、ある解析に特化したGalaxyも多数公開されています。ツールが絞られているかわりに、UseGalaxyには入っていないツールや独自に開発されたツールがインストールされています。以下がそのようなサイトの例です。
最近の動向としては、EBIがシングルセル向けのGalaxyツールを複数開発し下記のHuman Cell Atlas Galaxyに乗せています。
https://galaxyproject.org/use/human-cell-atlas/ (シングルセル解析に特化)
https://hicexplorer.usegalaxy.eu (Hi-C解析に特化)
http://huttenhower.sph.harvard.edu/galaxy/ (メタゲノム解析に特化)
公共Galaxyでできること、できないこと
多数のツールがインストールされた公共Galaxyをインターネット経由で使えるのは大変便利ですが、一方で制限もあります。たとえばusegalaxy.orgの場合のできることとできないことを見てみましょう。| できること | 制限 or できないこと |
|---|---|
| 900以上のツールがインストール済 レファレンスが多くの種から選べる 誰でも無料で利用可 公開データライブラリの利用 公開ヒストリー, 公開ワークフローの利用 ワークフローの作成 |
データライブラリの作成 ToolShedからのツール追加 レファレンスの追加 ユーザーあたり同時実行のジョブは6つまで ユーザーあたり使用可能なストレージは250GBまで |
ダウンロードしたばかりの素のGalaxyはこんな感じ
Galaxy本体をローカルにインストールすること自体はあまり難しいものではなく、開発元のインストール手順に書かれているミニマムなrequirementはOS(Unix/Linux or Mac OSX)とpython2.7のみです(※実際にはgitコマンドも必要です)。 https://galaxyproject.org/admin/get-galaxy/ しかし書いてあるとおりにインストールし、webブラウザからアクセスしてみると...。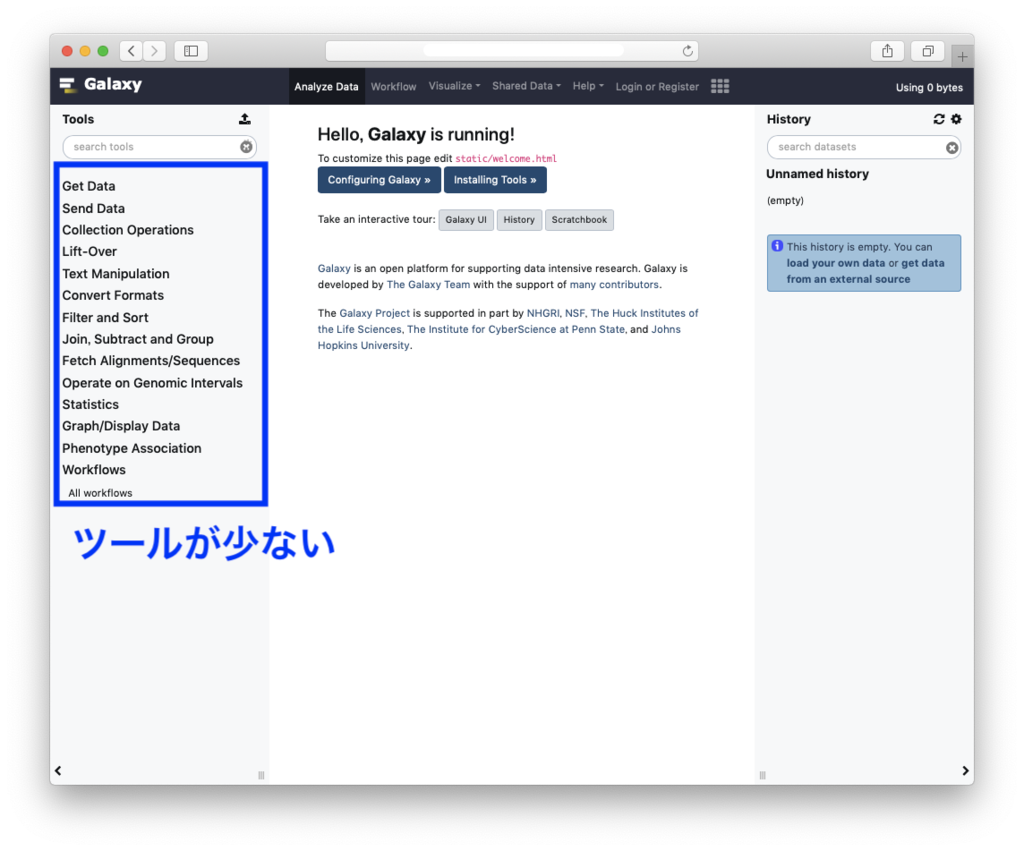 公共サーバーとは見た目からしてだいぶ違っています。ダウンロードした素のGalaxyは、ツールがとても少なく、レファレンスも設定されておらず、標準ではオフになっている機能が多くありミニマムな状態です。シングルユーザーが手元で試してみたい、という場合にはMacでも動作します。しかしwebサーバー、データベースサーバーとしての機能も最小限ですので、このままの状態だとチームで共有するための解析プラットフォームとしては実用的ではありません。なおDocker版も最初は同じようにミニマムな状態のGalaxyです。
逆に言えば、カスタマイズの自由度が高いということでもあります。前述した3つの地域別公共サーバー(UseGalaxy)を見比べてみても、デザインやインストールされているツールはそれぞれ違っています。自分専用のミニマムなものから、多数ユーザーがアクセスする公共サーバーまで使い方はさまざまです。カスタマイズに必要な材料は開発元から提供されていますので、ローカルにインストールしたGalaxyに対して目的や環境に合わせて必要な機能やツールを加えることで、理想の解析プラットフォームに近づいていくというわけです。
公共サーバーとは見た目からしてだいぶ違っています。ダウンロードした素のGalaxyは、ツールがとても少なく、レファレンスも設定されておらず、標準ではオフになっている機能が多くありミニマムな状態です。シングルユーザーが手元で試してみたい、という場合にはMacでも動作します。しかしwebサーバー、データベースサーバーとしての機能も最小限ですので、このままの状態だとチームで共有するための解析プラットフォームとしては実用的ではありません。なおDocker版も最初は同じようにミニマムな状態のGalaxyです。
逆に言えば、カスタマイズの自由度が高いということでもあります。前述した3つの地域別公共サーバー(UseGalaxy)を見比べてみても、デザインやインストールされているツールはそれぞれ違っています。自分専用のミニマムなものから、多数ユーザーがアクセスする公共サーバーまで使い方はさまざまです。カスタマイズに必要な材料は開発元から提供されていますので、ローカルにインストールしたGalaxyに対して目的や環境に合わせて必要な機能やツールを加えることで、理想の解析プラットフォームに近づいていくというわけです。
この状態から機能追加・ツールを揃えて構築していく
Galaxy導入にあたり考慮するポイントは- ユーザー数
- 解析内容(分野、どんなツールを使いたいか)
- ユーザーの範囲 (ラボ内だけか、共同研究者など外部からのアクセスがあるか)
- データの種類 (サイズ、数、新しいものが続々と来るか、同じものを繰り返して使うか、など)
- 使用パターン (ルーチンで同じツールを繰り返し実行するか、いろいろなツールをインタラクティブに使うか、など)
- ハードウェア環境 (計算ノードが何台もあるクラスターシステムか、ワークステーション1台か、など)
