前回までの記事
Galaxyをわかりたい!(1) Galaxyのキホン
最小限の状態のGalaxyをインストールすると、何かと”使ってみてはじめてわかる壁”に当たり、「ここをこうしたい」「こういうふうにできるのか」という希望が出てくると思います。そこで今回は、標準ではオフになっている拡張機能を目的ごとに紹介していきたいと思います。
ユーザーのアクセス制御をしたい
公開Galaxyは、誰でもアクセスして使うことができます。ローカルにインストールしたGalaxyも、Galaxyのホストと同じネットワーク内のユーザーならば同様に使用可能です。より厳密なアクセス管理をしたい場合、ユーザー登録の可不可や認証方法を変更することができます。
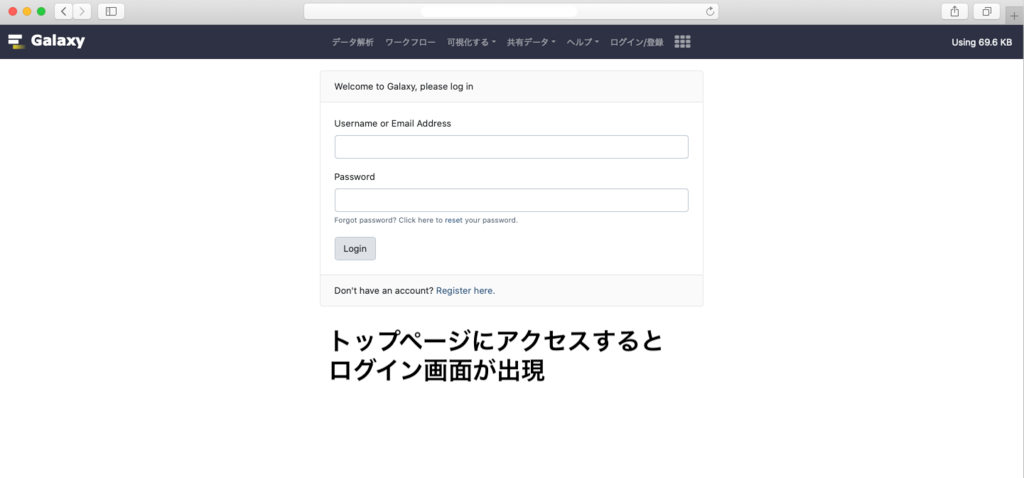
ログインしたユーザーのみが使用できるようにする
デフォルトでは、機能に制限はあるものの、ユーザー作成なしでもデータをアップロードして使用可能な状態になっています。これを、アカウントを作ったユーザーのみが使用可能なように設定できます。ユーザーはトップページへアクセスする前にログインが必要となります。
ユーザー作成を管理者のみが行うようにする
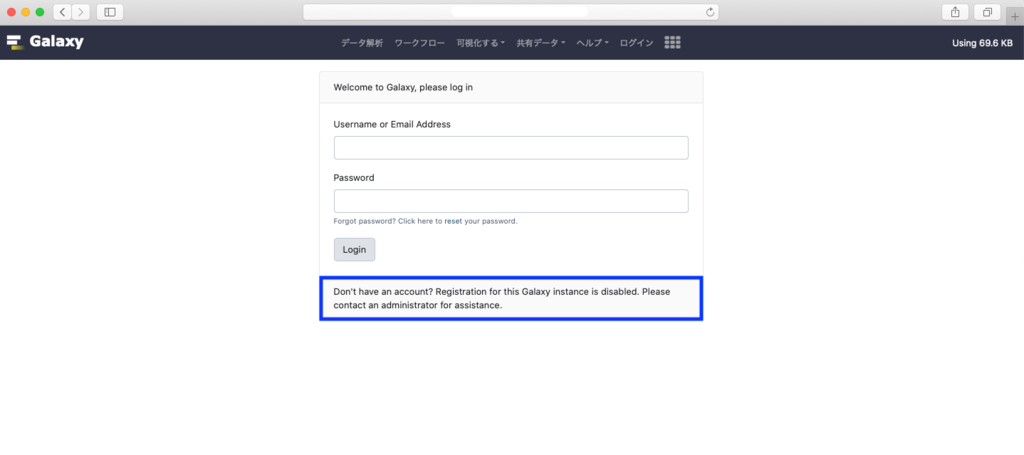
webインターフェースのログイン/登録からユーザー自身が登録することができるようになっていますが、これを許可せずに管理者による登録のみに限定することができます。前述のログインユーザーのみ使用の設定と合わせることで、管理者が許可したユーザーのみがGalaxyにアクセス可能となります。
デフォルトのログイン画面では、ユーザー作成のためのリンクが表示されていますが、管理者のみがユーザー作成を行う設定になっていると、ユーザー作成画面へのリンクは出現せず、管理者にコンタクトするよう促すメッセージが表示されます。
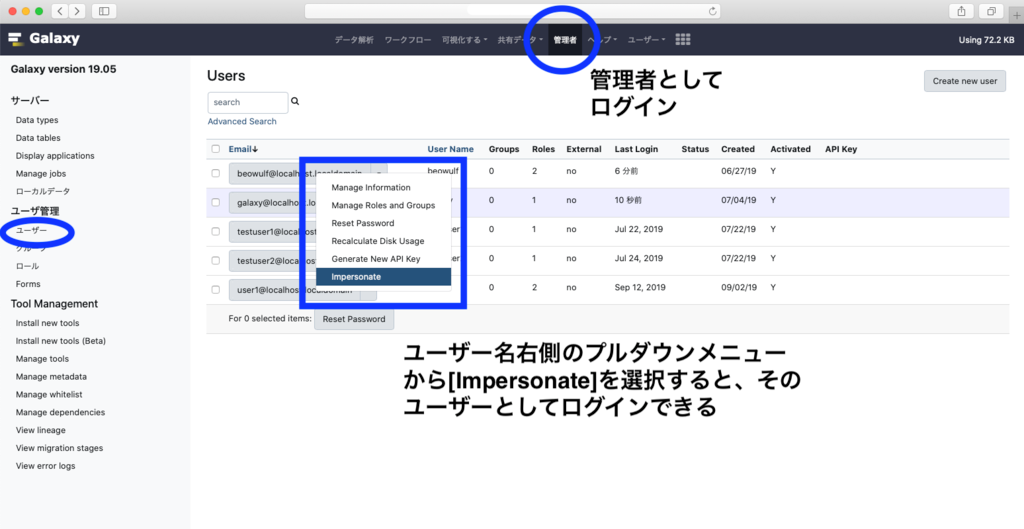
管理者が代理ログインできるようにする
管理者が別のユーザーとしてログインできるようにするかどうかの設定ができます。ユーザーのサポートやエラー解析の際に便利です。
外部の認証ツールと連携させる
Galaxyで作成したユーザー情報はGalaxyのデータベースに格納されますが、LDAP ,Active Directory, Apacheの認証プロキシといった外部の認証ツールと連携させることができます。たとえばLDAPで使用しているものと共通のユーザー名とパスワードでGalaxyにログインするといったことが可能です。
サイズの大きいデータも含め、データのアップロード方法を増やしたい
Galaxyにデータをアップロードする場合、webUIでクライアントPCのデータを選んでアップロードするのが最も簡単な方法です。しかしこの方法はサイズに2GBの制限があります。2GB以上のデータを扱うための方法を2つご紹介します。
SFTP,FTPSを使ったファイルのアップロード
まず1つ目はFTPアップロードです。暗号化されていないFTPプロトコルはセキュリティ上の問題がありますが、より安全なSFTP,FTPSを使うことができます。ユーザー名とパスワードはGalaxyのユーザーアカウントを使用するので、Galaxyが動作しているホストのアカウントがなくてもアップロードできます。公開Galaxyではこの方法でアップロードが可能ですが(FTPSのみ対応)、ローカルにインストールしたGalaxyでは標準では使用できません。以下の条件を満たすことで使用可能となります。
- Galaxyが動作しているホストでSFTP/FTPSに対応したFTPサーバーが動作している。
- FTPサーバーが認証時にGalaxyのユーザーアカウントを使用するよう設定されている。
- Galaxy側でFTPアップロードが有効となっている。
- ユーザーのクライアントPCでSFTP/FTPSに対応したFTPクライアントが使用可能である。
FTPクライアントとしてよく使われるFFFTP、WinSCP、Cyberduck、FIlezillaは、SFTPかFTPS、あるいはその両方に対応していますし、MacのターミナルやWindowsのPower ShellではSFTPコマンドが標準で使えます。
データライブラリ(Shared Data)
2つ目はデータライブラリです。チーム内で共有するデータの場合は、管理者があらかじめデータライブラリを作成してそこにアップロードしておくのも手です。下の画像は、データライブラリ管理メニューです。

ユーザーはwebUIの共有データ(Shared Data)メニューからデータライブラリのデータを自分のヒストリーにインポートすることができます。データライブラリの作成は管理者権限が必要なため、開発元の公開Galaxyでは新規作成ができません。自前でGalaxyを構築することで使えるようになる機能です。
たとえばラボの実験データや変異解析のデータベースをデータライブラリにしておくことで、個々のユーザーが大きいデータをアップロードする手間が省けます。また、Galaxyにおいてユーザーごとにディスク容量などのリソース制限を設定している場合、データライブラリの分は含まれないのでその点もメリットと言えるでしょう。
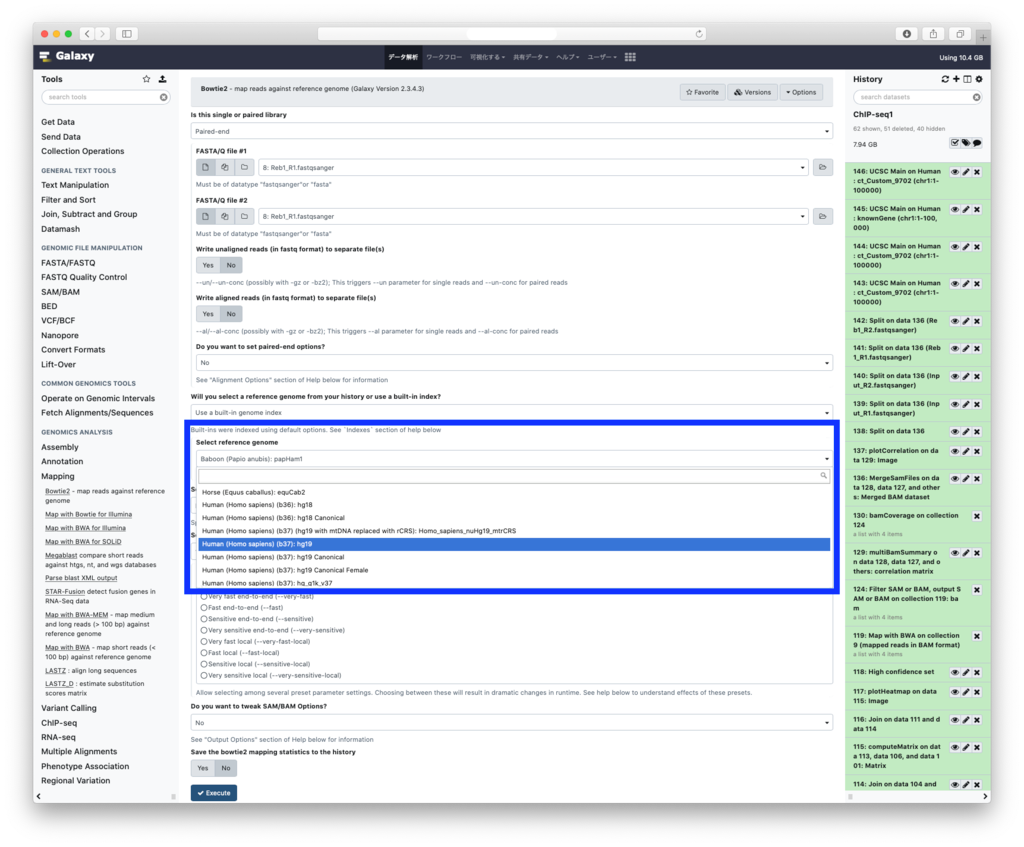
いろいろな種のレファレンスデータを使えるようにしたい
Bowtie2,BWAといったマッピングツールを使うときはゲノムレファレンスデータが必要です。Galaxyでは
- ツールのプルダウンメニューからゲノム種を選んで使用
- ユーザーが自分でヒストリーにアップロードしたゲノムデータからインデックスを作成して使用
と2つの方法があります。ツールから選べるようにするには、管理者があらかじめツールごとに設定しておく必要があり、標準ではレファレンスゲノムは何も入っていません。Galaxyが動作しているホストにレファレンスインデックスを配置し、Galaxyの設定ファイルに追加する必要があります。しかし数種類程度ならさほど問題にならないこの作業も、レファレンスデータを用意するところから始めないといけませんし、たくさんのゲノム種を設定するには手間暇がかかります。GalaxyではCVMFSを使ってインターネット経由で自動的にダウンロードする方法もあります。
CVMFSを使ったゲノムデータ取得
CVMFS(CERN VIrtual Machine File System)はインターネット越しに共有できるread-onlyのファイルシステムで、ソフトウェア配布のためによく使われます。公開GalaxyのレファレンスデータはCVMFSで使用可能となっており、ローカルのGalaxyからマウントして同じデータ群を取得可能です。インターネット経由なので初回のダウンロードには時間がかかりますが、管理者がひとつひとつ設定しなくても最初から公開サーバーと同じレファレンスデータを選ぶことができます。ただし、全てのツールに対応できているわけではありませんので、ツールによっては従来通り個別に設定する必要が出てきます。
Galaxyの処理能力を上げたい
Galaxy自体のインストールはシンプルで、git cloneでダウンロードしてシェルスクリプトを実行するだけで起動します。webサーバーとデータベース、ジョブマネージャーはビルトインのものが使用されており、設定を意識しなくてもすぐにアクセスすることができます(最初の最初はlocalhostのみですが)。それだけならパソコンでも動作するくらいに軽いのですが、ユーザーやデータが増えて実運用となるとやはりそれだけでは不足です。Galaxyにはプロダクション環境での運用に対応してカスタマイズ可能になっている部分がいくつかありますのでご紹介します。いずれもGalaxy本体と外部ツールとの連携ですので、管理者がGalaxyホストにログインしてコマンドラインで外部ツールのインストール・設定を行う必要があります。
Webサーバ
ApacheもしくはNGINXを別に立ててそれをwebサーバーとすることができます。リバースプロキシの機能を持たせたいときに有用です。IGVにデータを転送して表示させるにはビルトインのwebサーバーでは実現できず、外部webサーバーの設定が必要となります。
データベース
GalaxyはSQLデータベースにユーザー情報やデータ、ヒストリー、ツールのIDなどのメタデータを格納します。デフォルトではsqliteを使用しますが、PostgreSQL,MySQLに変更することができます。社内でテストしたところでは、デフォルトのsqlite使用だと連続でジョブを投入した際にGalaxyの挙動が不安定になることがありましたが、PostgreSQLに変更してからはその心配がなくなりました、MySQLでも同様です。
ジョブスケジューラ
バックエンドのマシンがクラスター構成の場合、Galaxyから実行したジョブをUniva Grid Engine, Slurmなどのジョブスケジューラで実行させることができます。どの計算ノードで実行されるかをユーザーが意識することなく、ジョブを分散させることができます。ツールごとにあらかじめ使用キューを指定しておいたり、マルチスレッド実行を設定したりもできます。
Pulsarでリモートシステムにジョブを投入
Pulsarはリモートシステムでジョブを実行するために開発されたGalaxy専用のツールです。入力ファイルやジョブの実行に必要なconfigファイルもリモートシステムに転送されるので、NFSで共有している必要がありません。クラウドでジョブを実行させることもできます。
Galaxyツールを自分で開発したい
Galaxy Tool ShedではコミュニティベースでたくさんのGalaxyツールが開発されアップロードされています。しかし「まずコマンドラインのアプリケーションの開発があり、それを有志がGalaxyに適用させる」という順序となることがほとんどのため、新しい手法のアプリケーションや最新バージョンはどうしても一歩遅れがちになります。また、既存のGalaxyツールではアプリケーションの全てがコマンドラインと同様に使えるとは限りません。ツールの作り方によってはサブコマンドやオプションが省かれるということも往々にしてあることです。そのような場合はいっそ自分でツールを作ってしまいたいということになるでしょう。
Planemo
PlanemoはGalaxyが提供しているツール開発のためのユーティリティです。xml形式の設定ファイル、テストデータなど、Galaxyツールの部品を生成やテスト、ToolShedへの公開など継続した開発をこのPlanemoで行うことができます。最近のバージョンはCWLなど、開発中の新しいGalaxyツール実装方法にも対応しているようです。
Galaxyをコマンドラインで操作したい
webブラウザからGUIで操作できるのがGalaxyの最大のメリットではあります。しかし、管理する立場で何度も同じ操作をするようなケース、GUIの解析ツールで細かいオプションを指定したい場合はコマンドラインで操作したくなる時もあります。そのような要求に対応できる機能をご紹介します。Galaxy管理をコマンドラインで行えるのがBioblend、解析をコマンドライン(PythonもしくはR)で実行できるのがInteractive Environmentです。
Bioblend
Galaxyでは、pythonのAPIライブラリBioBlendを使うと自由度の高い操作ができます。たとえばデータライブラリ(Shared Data)の場合、管理者でログインするとライブラリの新規作成やデータアップロードをwebUIからすべて行うことができますが、以下はBioblendを使ってライブラリ新規作成とファイルアップロードの部分を書いた例です。ログインするかわりに、APIキーを使います。
from bioblend.galaxy import GalaxyInstance
gi=GalaxyInstance(utl="http://localhost:8083", key=myAPIkey)
role_id=gi.roles.get_roles()[5]['id']
new_lib=gi;libraries.create_library('shared_fastqdata')
gi_lib_set=gi.libraries.set_library_permissions(net_lib['id'], access_in=[role_id],
modify_in=[role_id], add_in=[role_id], manage_in=[role_id])
datalist=gi.libraries.upload_file_from_server(new_lib['id'], 'fastqdata')
Interactive Environment (Jupyter, RStudio)
Galaxyのツール群はプルダウンメニューやwebフォームで入力パラメータを指定できます。しかしデータのフィルタリングや図表作成など、条件を少しずつ変えて結果を確認しながら作業したい時には、GUIのツールではどうしても細かい調整ができないのが難点です。GalaxyはJupyterあるいはRStudioと連携する機能を提供しており、Galaxyのパネル上でJupyter/RStudioを起動して操作することができます。すなわち、Galaxy上でPythonもしくはRのコードを書いて実行できるのです。またGalaxyデータを取り込むインターフェースが用意されているので、GalaxyヒストリーのデータをJupyter/RStudioにインポート/エクスポートしたり、履歴(notebook)をGalaxyヒストリーにインポート/エクスポートすることができます。
このInteractive Environmentは、一つのトピックとして後の回で詳しくご説明したいと思います。



 ユーザーはwebUIの共有データ(Shared Data)メニューからデータライブラリのデータを自分のヒストリーにインポートすることができます。データライブラリの作成は管理者権限が必要なため、開発元の公開Galaxyでは新規作成ができません。自前でGalaxyを構築することで使えるようになる機能です。
たとえばラボの実験データや変異解析のデータベースをデータライブラリにしておくことで、個々のユーザーが大きいデータをアップロードする手間が省けます。また、Galaxyにおいてユーザーごとにディスク容量などのリソース制限を設定している場合、データライブラリの分は含まれないのでその点もメリットと言えるでしょう。
ユーザーはwebUIの共有データ(Shared Data)メニューからデータライブラリのデータを自分のヒストリーにインポートすることができます。データライブラリの作成は管理者権限が必要なため、開発元の公開Galaxyでは新規作成ができません。自前でGalaxyを構築することで使えるようになる機能です。
たとえばラボの実験データや変異解析のデータベースをデータライブラリにしておくことで、個々のユーザーが大きいデータをアップロードする手間が省けます。また、Galaxyにおいてユーザーごとにディスク容量などのリソース制限を設定している場合、データライブラリの分は含まれないのでその点もメリットと言えるでしょう。


